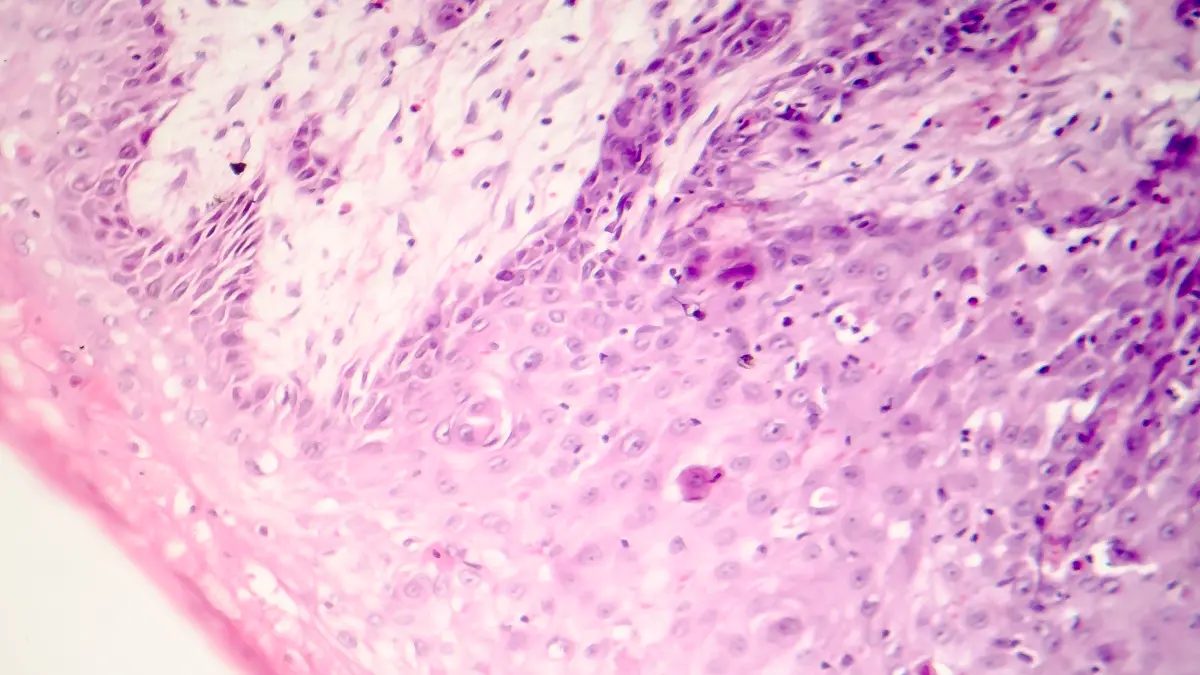
Ein KI-Modell aus Dresden erkennt in Gewebeproben mehrere genetische Darmkrebs-Merkmale gleichzeitig. Das kann Diagnostik beschleunigen und gezielte Therapien sowie Vorscreenings unterstützen.
Die multizentrische Studie analysierte fast 2.000 digitalisierte Gewebeschnitte von Patientinnen und Patienten mit Darmkrebs aus sieben unabhängigen Kohorten in Europa und den USA. Die Proben umfassten sowohl Schnittbilder von Gewebeproben als auch klinische, demografische und lebensstilbezogene Daten. Die Forschenden entwickelten ein neuartiges „Multi-Target-Transformer-Modell“, das eine Vielzahl genetischer Veränderungen direkt aus standardmäßig histologisch gefärbten Gewebeschnitten von Darmkrebsproben vorhersagen kann. Frühere Studien waren meist darauf beschränkt, jeweils nur eine genetische Veränderung vorherzusagen – gleichzeitig auftretende Mutationen sowie die damit einhergehenden Veränderungen im Gewebe blieben weitgehend unberücksichtigt.
„Bisherige Deep-Learning-Modelle und die Analyse der zugrunde liegenden Gewebeveränderungen beziehen sich in der Regel auf nur jeweils eine Mutation. Unser neues Modell kann viele Biomarker gleichzeitig identifizieren, so auch bislang klinisch nicht beachtete. Das konnten wir in mehreren unabhängigen Kohorten nachweisen. Dabei haben wir festgestellt, dass viele Mutationen gehäuft in mikrosatelliteninstabilen (MSI) Tumoren auftreten,“ erklärt Marco Gustav, Erstautor der Studie und Wissenschaftler am EKFZ für Digitale Gesundheit der TUD. Gewisse Arten von Darmkrebs können anhand der Mikrosatelliteninstabilität (MSI) klassifiziert werden. Mikrosatelliten sind kurze, sich wiederholende DNA-Sequenzen, die über das gesamte Genom verteilt sind. Bei Krebs kann es zur MSI kommen, wenn diese Sequenzen infolge eines Defekts im DNA-Reparatursystem instabil werden. MSI dient als wichtiger Biomarker, um Patientinnen und Patienten zu identifizieren, die von einer Immuntherapie profitieren könnten. „Das deutet darauf hin, dass verschiedene Mutationen zusammen zum veränderten Erscheinungsbild des Gewebes beitragen. Das Modell erkennt also gemeinsame visuelle Muster – anstatt genetische Veränderungen unabhängig voneinander zu identifizieren“, fügt er hinzu.
Die Forschenden zeigten, dass ihr Modell bei der Vorhersage zahlreicher Biomarker wie BRAF- oder RNF43-Mutationen und MSI direkt aus pathologischen Präparaten genauso gut abschnitt wie bereits etablierte Modelle – und manche von ihnen sogar übertraf. Die pathologische Expertise zur Beurteilung von Gewebeveränderungen anhand histologischer Schnitte wurde von erfahrenen Medizinerinnen und Medizinern eingebracht. Dr. Nic Reitsam vom Universitätsklinikum Augsburg war dabei maßgeblich an der Studie beteiligt.
Zur Bedeutung der Studie sagt Jakob N. Kather, Professor für Klinische Künstliche Intelligenz am EKFZ für Digitale Gesundheit der TUD und leitender Onkologe am NCT/UCC des Universitätsklinikums Carl Gustav Carus Dresden: „Unsere Forschung zeigt, dass KI-Modelle diagnostische Abläufe deutlich beschleunigen können. Gleichzeitig gewinnen wir mit diesen Methoden neue Einblicke in den Zusammenhang zwischen molekularen und morphologischen Veränderungen bei Darmkrebs. Künftig könnte diese Technologie als effektives Vorscreening-Werkzeug zum Einsatz kommen – und Ärztinnen und Ärzte dabei unterstützen, gezielt Patientinnen und Patienten für weiterführende molekulare Tests auszuwählen und individuelle Therapieentscheidungen zu treffen.“
Derzeit plant das Forschungsteam, diesen Ansatz auch auf andere Krebsarten zu übertragen.
Die Studie entstand in interdisziplinärer Zusammenarbeit zahlreicher Wissenschaftlerinnen und Wissenschaftler an führenden Forschungseinrichtungen in Europa und den USA. Neben der TUD und dem Dresdner Universitätsklinikum waren u. a. die Medizinische Fakultät der Universität Augsburg, das Nationale Centrum für Tumorerkrankungen (NCT) in Heidelberg, das Fred Hutchinson Cancer Center in Seattle (USA), die Medizinische Universität Wien (Österreich) und die Mayo Clinic (USA) beteiligt.
Originalpubliaktion: Gustav M, van Treeck M, Reitsam NG et al: Assessing Genotype-Phenotype Correlations with Deep Learning in Colorectal Cancer: A Multi-Centric Study. The Lancet Digital Health, 2025
Quelle: Pressemitteilung Technische Universität Dresden
Bilderquelle: © Anja Stübner / EKFZ